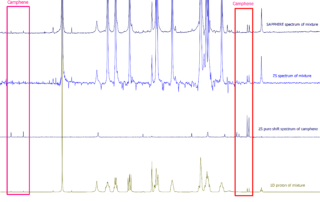
JASON 4.1が登場
JASON 4.1が登場 今回のリリースで、JASON 4.1はWindowsとmacOSの両方で「ダークテーマ」をサポートします。お使いのOSをダークモードに切り替えるだけで、JASONがダークになります! スペクトルの描画品質、ベクターグラフィックスおよびビットマップ出力に多くの改良を加えました。また、macOSでの2D等高線プロットの応答性を向上させました。さらに、macOSでのNUS処理を高速化しました。 パルスシーケンスのソースコードを、テキスト文書としてNMRパラメータとともに表示できます。 Zipアーカイブはキャンバスにドラッグ&ドロップするだけです。JASONは、開くことができるすべてのファイルを自動的に解凍します! 分子内の原子にはカスタムラベルを付けることができます。こうすることで、必要に応じて原子の 「番号変更」や「マーク付け」を行うことができようになりました。 SMILEQをさらに高速化し、レポートエディターを追加しました。 [...]